事例3
Mass++を用いたLC-MALDI-TOF/TOFデータの解析例
2014-3-4 東京都健康長寿医療センター 老化機構研究チーム 研究員 津元 裕樹
(http://www.tmghig.jp/J_TMIG/kenkyu/team/proteome.html)
我々の研究チームではnanoLC-MALDI-TOF/TOFシステムを用いてプロテオーム解析を行っています。今回、Mass++を用いてデータを読み込めるように対応して頂き、Heatmap表示機能を用いてデータを見ることでデータベース検索だけではわからなかった翻訳後修飾が存在することがわかってきましたので紹介します。
あるタンパク質のトリプシン消化物を分析したところ、異なる保持時間で同じm/z値を有するシグナルAおよびBから同じペプチドが同定されました(Fig 1)。しかしながら、同じペプチドが異なる保持時間で同定された理由については不明でした。
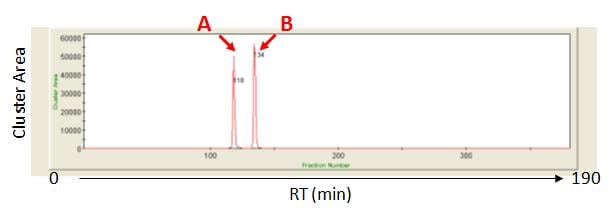
Fig1. 抽出イオンクロマトグラム
そこで、データをmzML形式に変換してMass++で読み込み、Heatmap表示を行いました(Fig 2)。その結果、赤矢印で示すように、シグナルAおよびBに対して高分子量側へシフトするシグナルが存在すること、早い保持時間で検出されたシグナルAについては、さらに高分子量側にシグナルCおよびDが存在することが分かりました。これら赤矢印で示したm/z値のMS/MSスペクトルはすべて同じパターンであったことから同じ配列のペプチドに対する翻訳後修飾であり、シグナルCおよびDについてはその分子量差から酸性の翻訳後修飾であると考えています。今回の解析結果は新たな翻訳後修飾の可能性を示すものであり、研究の発展が期待されます。
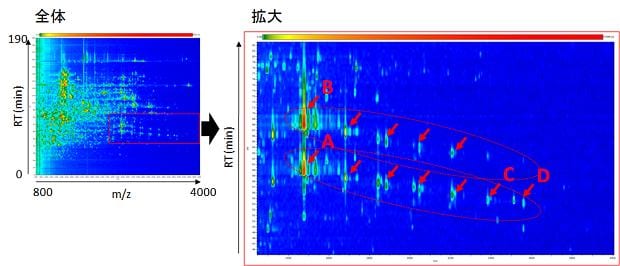
Fig2. Mass++を用いたLC-MALDIデータのHeatmap表示
Mass++のHeatmap表示機能は見やすく、シグナルの違いを見分けるには有用でした。今後、LC-MALDIデータの解析ソフトとしてさらに開発が進むことを期待しています。


